Nat Commun:赋予单细胞精确空间坐标!索生宝/彭广敦团队创新开发高分辨空间单细胞映射方法CMAP
2025-07-19 测序中国 测序中国 发表于上海
CMAP 方法整合单细胞与空间转录组数据,实现单细胞精确空间定位,适应性强,助力解析组织微环境与癌症免疫等复杂信息。
近年来,虽然单细胞RNA测序(scRNA-seq)在肿瘤学、神经学、发育生物学、免疫学等多个医疗领域实现了突破性应用,但由于忽略了细胞的空间信息,无法了解细胞外和细胞内相互作用在组织环境中的关键作用。空间转录组学(ST)技术的出现,为解决这一难题提供了有力的工具,但存在低捕获率、转录组覆盖不完整,以及无法准确分割细胞等限制。因此,空间分析的下游应用需要真正的单细胞分辨率和原位组织的灵敏度。
为此,广州国家实验室索生宝团队联合中国科学院彭广敦团队提出了一种从ST数据重建空间细胞剖面的创新方法CMAP(Cell Mapping of Attributes with Position)。该方法通过整合单细胞和空间转录组数据,可以有效地将大规模单细胞映射到其精确空间位置,重建全基因组空间基因表达谱,解锁了以增强单细胞分辨率探索组织微环境的潜力。经模拟和真实数据集的分析表明,CMAP在不同数据类型和测序平台上都具有良好的适应性。特别是在scRNA-seq和ST数据不匹配的情况下,CMAP表现依然良好,可实现可靠的集成和解释。

CMAP提供了一个强大的、兼容的和可扩展的方法来绘制单细胞分辨率的空间细胞图谱,其赋予单细胞精确空间坐标的能力有助于解析细微的空间器官特异性内皮细胞异质性,以及传统单细胞或空间数据分析无法捕捉的复杂癌症免疫微环境信息。
CMAP概述
研究团队以Visium数据为例概述了CMAP工作流程。CMAP通过从空间域逐步进展到检测的空间spot,系统地绘制细胞图谱,最终获得精确的空间位置(图1a)。该流程由三个主要过程组成:CMAP-DomainDivision(一级映射)将细胞划分到空间域,CMAP-OptimalSpot(二级映射)将细胞对齐到最佳spot,CMAP-PreciseLocation(三级映射)确定精确的细胞坐标。
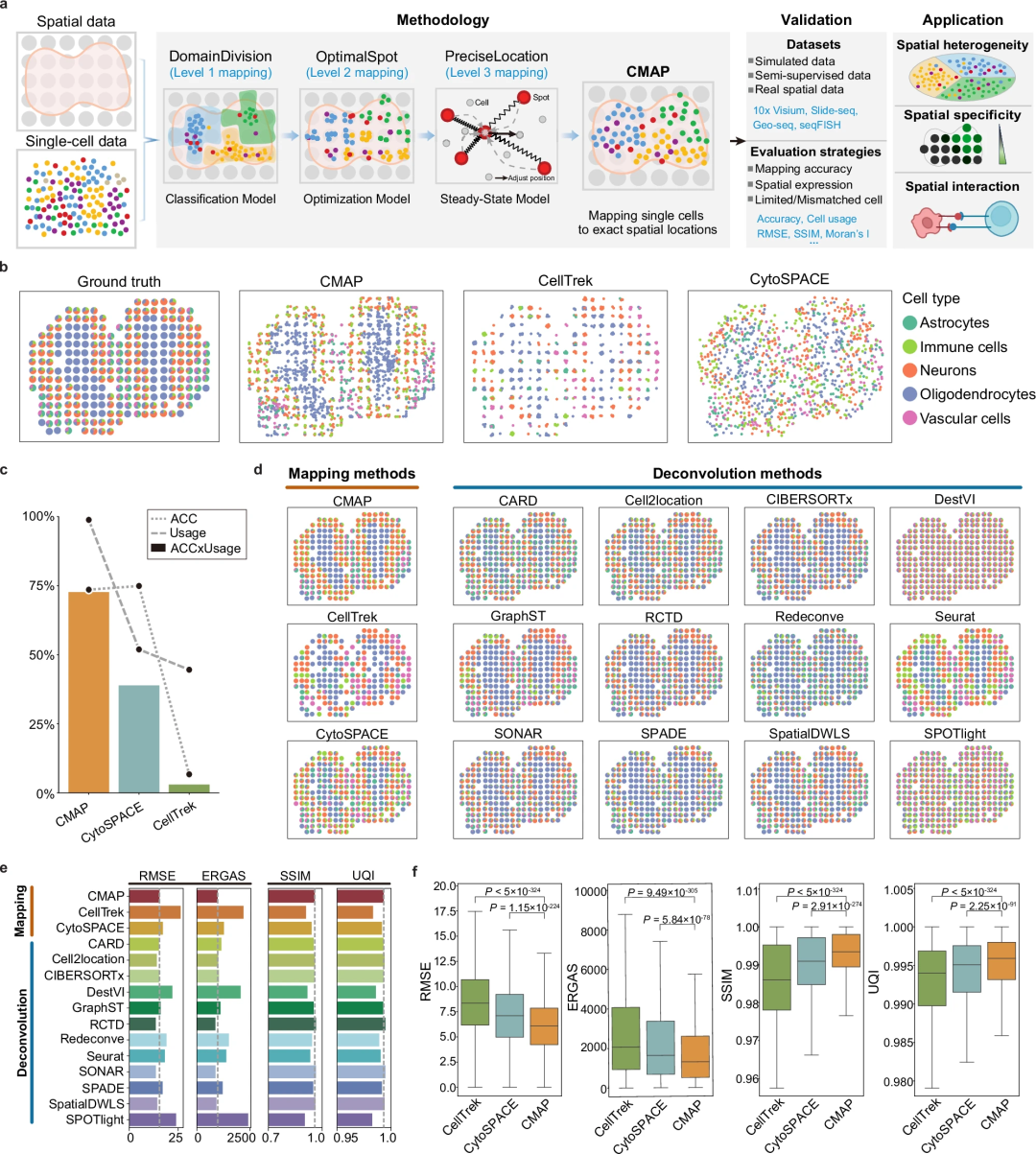
图1.CMAP示意图和性能评估。
在CMAP-DomainDivision中,研究团队利用ST数据中的表达谱和空间坐标来识别空间特异性基因,并使用隐马尔可夫随机场对空间域进行聚类。同时,CMAP也可以结合其他方法预定义的空间簇及其对应的特定领域基因。此外,研究团队训练了一个分类模型,来以最高的预测概率为单个细胞分配空间域标签,通过可调的概率阈值,方便去除与空间组织不匹配的细胞,并基于空间域内细胞类型分布进行先验的可行性评估。该过程中,CMAP通过预先选择和分配细胞到特定的空间域,大大减少了细胞映射的搜索空间,提高了计算效率,并揭示了空间异质性和细胞内类型的变化,这是无法仅通过单细胞RNA表达识别的。
CMAP-OptimalSpot中,在每个空间域中识别空间可变的基因,并生成细胞和spot之间的随机比对矩阵。与每个spot相连的细胞被聚类,并分析实际和聚合的空间表达模式之间的差异。研究团队采用了基于图像的结构相似性指数进行模式比较,以增强空间特征捕获和结构相似性评估,并进一步评估空间中的细胞分布密度。通过基于深度学习的优化迭代改进,CMAP得到连接细胞到各自spot的最优映射矩阵。
在CMAP-PreciseLocation中,研究团队首先构建了一个最近邻图来表示所有spot之间的关系。考虑到细胞与周围环境的相互作用,研究计算了细胞与邻近最佳spot之间的关联。并利用Spring稳态模型在该空间环境中为每个细胞分配确切的位置。值得注意的是,CMAP预测的细胞位置获得了超越单纯spot水平的精细坐标,有效地弥合了相邻spot之间的差距,并增强了空间分辨率。
性能评估
研究团队通过2个模拟数据集、1个高分辨率Xenium数据集和5个真实数据集全面评估了CMAP在性能,包括其在各种单细胞测序平台(例如Smart-seq2, 10x Genomics Chromium等)和空间技术平台(例如seqFISH、10x Genomics Xenium、Slide-seq、10x Genomics Visium和Geo-seq)上将细胞映射到其原生位置的强大能力,以证明其在空间解析单细胞方面的有效性和实用性,并使用最先进的方法(包括CellTrek、CytoSPACE和几种反卷积算法等)对其进行基准测试。
模拟基准测试结果显示,CMAP的性能优于CellTrek和CytoSPACE(图1,2)。CMAP绘制的细胞与Xenium数据之间的相对高度一致性强调了其捕获复杂空间组织的能力(图3)。在Xenium综合数据的基础上,系统考察关键参数对图谱绘制结果的影响,进一步凸显了CMAP的一致性和适应性。通过仔细评估空间域数量、批次效应去除和分类器选择等关键因素的影响,研究团队证明了CMAP可以在不同的实验设置中提供更可靠的映射,提供高保真的单细胞和空间数据的集成分析。
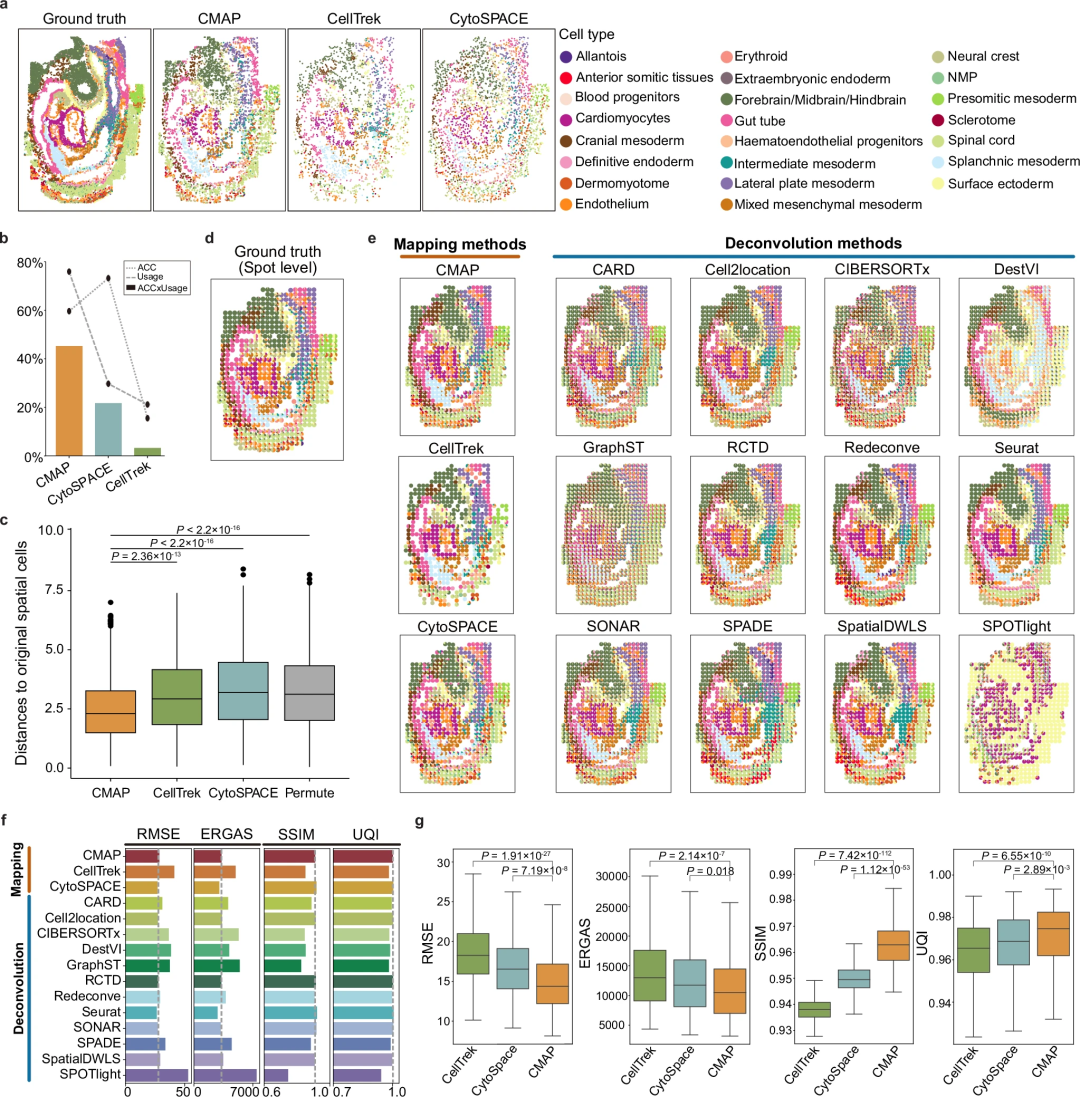
图2.基于模拟数据的CMAP评估。
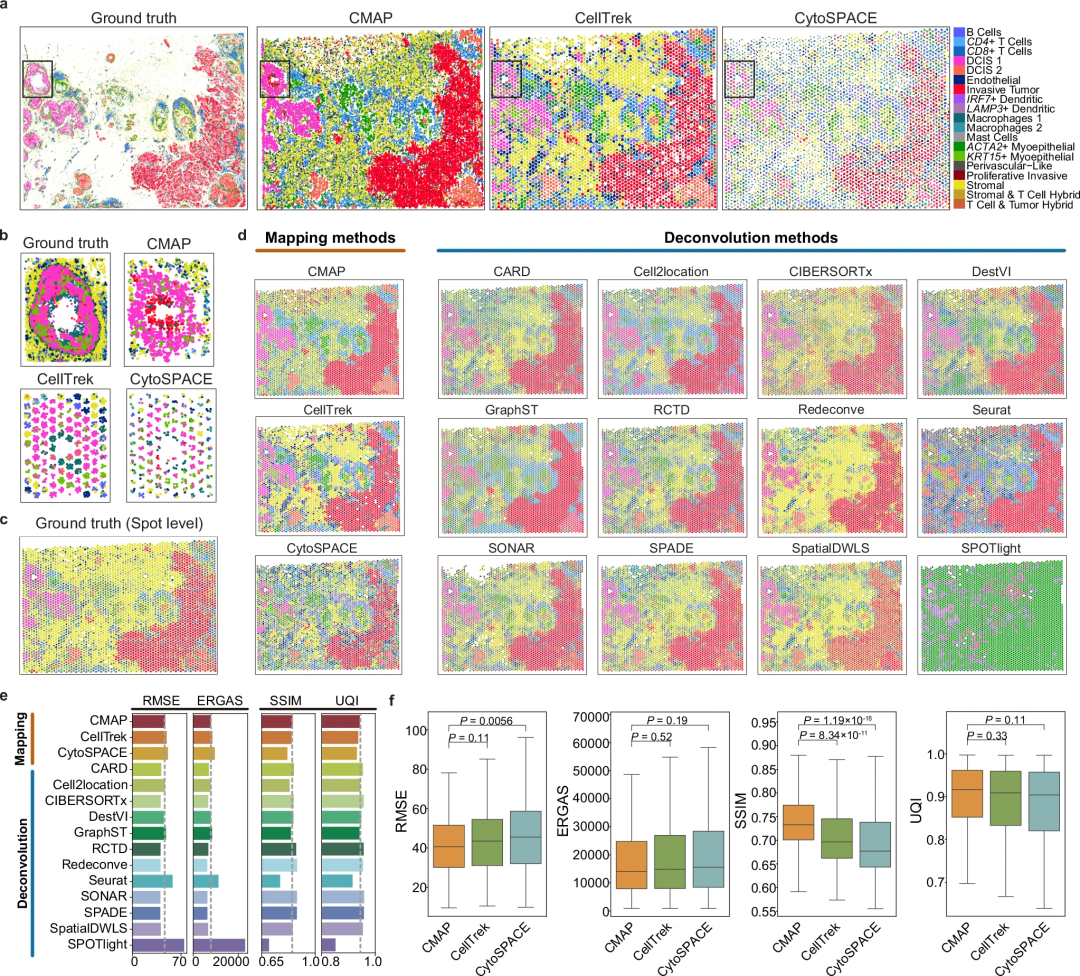
图3.CMAP在真实高分辨率Xenium数据(Xenium数据与Visium数据集成)上的评估。
在小鼠皮质数据与Visium空间数据的半监督数据集评估中,CMAP在将细胞定位到正确的解剖层方面显示出良好的准确性,展示了高精度处理小细胞群的能力;在涉及不匹配数据集的场景中,CMAP可区分并排除不匹配的细胞,确保只绘制相关的细胞群,从而提高空间图谱的可靠性(图4)。此外,在内皮细胞的应用中,CMAP证明了其在揭示细胞类型的空间异质性方面的实用性。
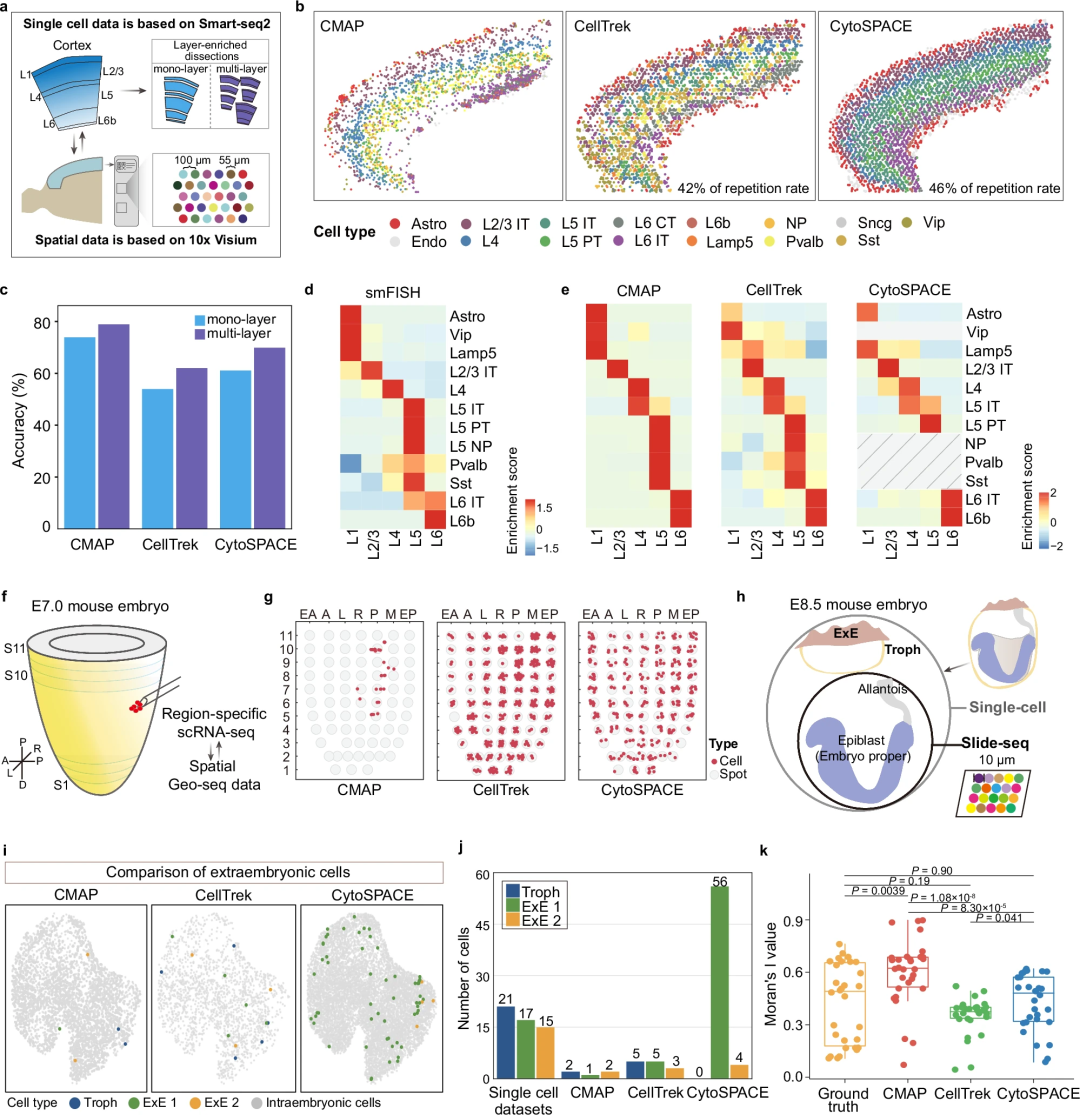
图4.CMAP在多种真实世界场景中的性能。
结语
CMAP通过整合scRNA-seq和ST数据来预测全面转录组覆盖的精确单细胞位置,实现了真正的单细胞分辨率,确保了整个转录组空间组织的准确重建。CMAP的灵活性允许其应用于各种场景,包括不匹配的细胞过滤、小细胞群定位和肿瘤微环境分析,有助于深入了解复杂组织中的细胞功能。未来,研究团队计划CMAP可以通过整合单细胞和空间维度的多组学数据,包括蛋白质组学和表观基因组学数据等,以提供对细胞空间异质性更全面的理解,揭示细胞功能和组织的重要调控机制。
原文信息:
Ke, J., Xu, J., Liu, J. et al. High-resolution mapping of single cells in spatial context. Nat Commun 16, 6533 (2025). https://doi.org/10.1038/s41467-025-61667-4
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言















#空间转录组# #CMAP#
6 举报