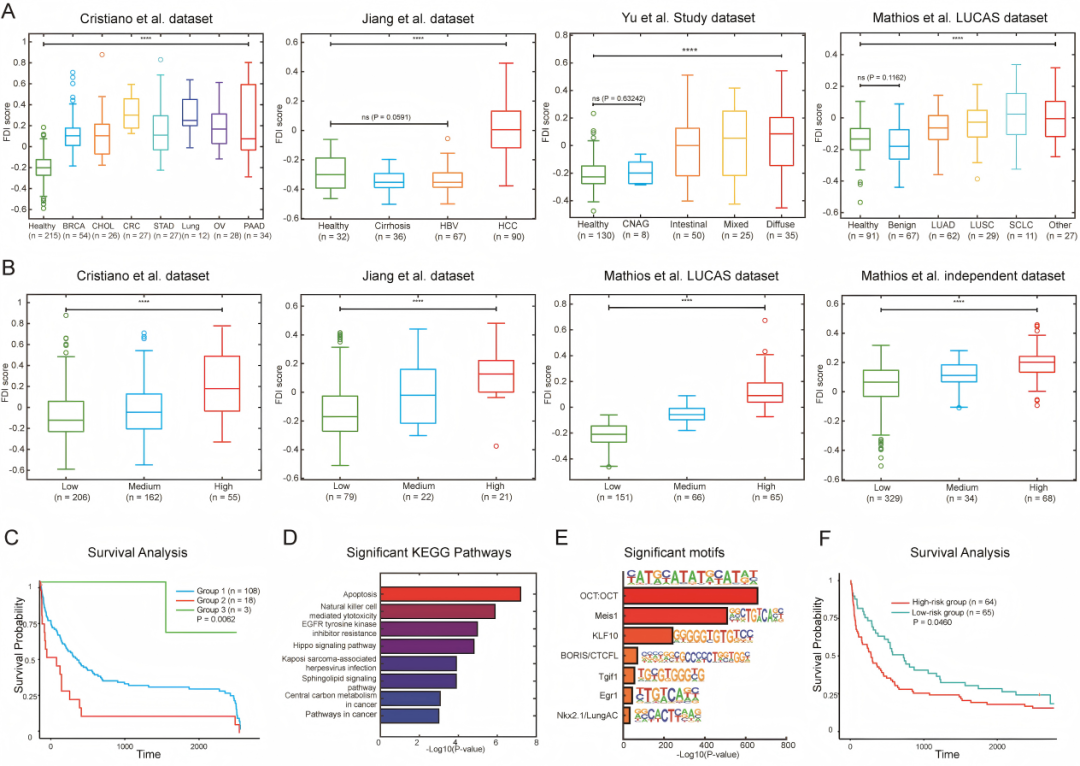
Cell Reports Methods:新型cfDNA片段模式FDI揭示染色质可及性,助力癌症早筛
2025-06-21 测序中国 测序中国 发表于上海
华中农大与军事医学科学院团队提出片段离散度(FDI)指标,可刻画染色质可及性,基于此构建的 FDI-Oncology-Model 能提升癌症早期检测能力,且在多个独立癌症队列中表现出稳健的诊断性能。
导读
近日,华中农大章文/周雄辉和军事医学科学院陈河兵团队在Cell Reports Methods期刊发表题为《Fragment dispersity index analysis of cfDNA fragments reveals chromatin accessibility and enables early cancer detection》的文章。该研究提出了一种融合cfDNA覆盖度变化与片段末端分布的片段模式:片段离散度(FDI),能够高效刻画染色质可及性区域,以此构建的FDI-Oncology-Model可以显著提升癌症早期检测的能力,为cfDNA片段组学在癌症筛查中的转化应用探索出一条可行的新路径。
研究发现
一、片段离散度可以刻画染色质可及性
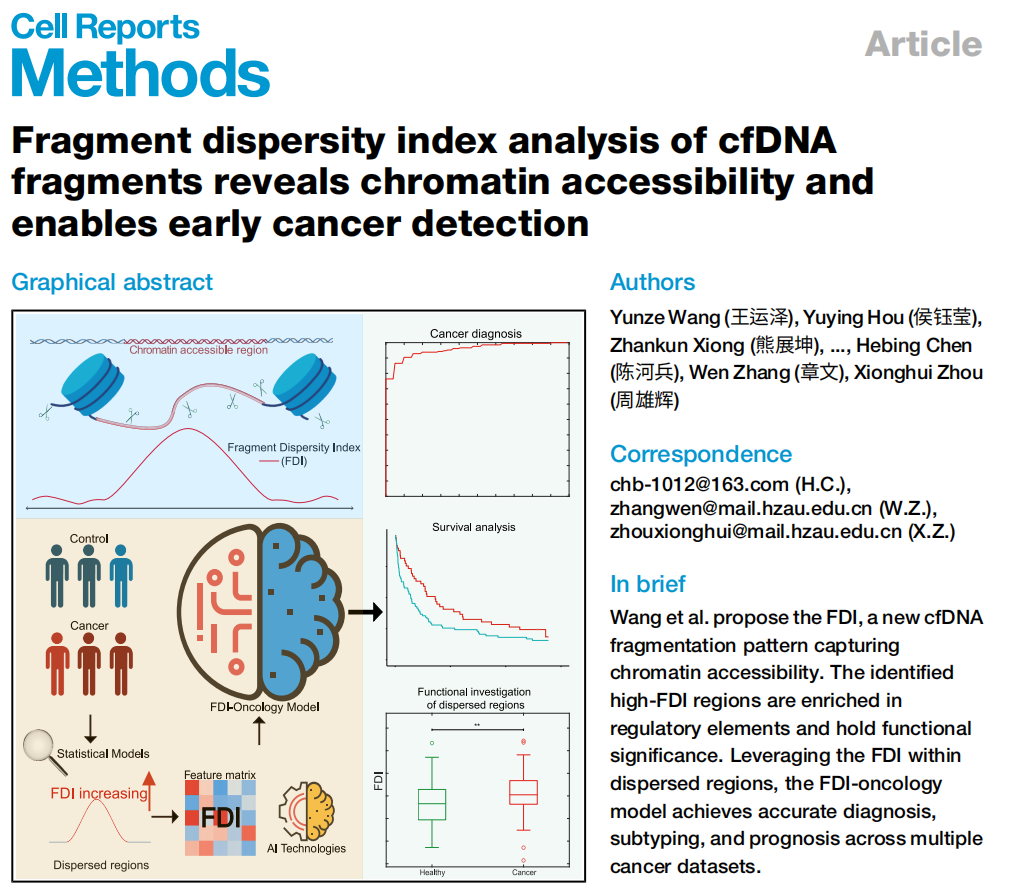
现有片段模式多从单一角度设计特征公式,对cfDNA中结构信息的利用仍有提升空间。该研究从片段终点分布与覆盖度变化的联合视角出发,提出了片段离散度(FDI)指标,用于更精准刻画染色质可及性区域。
该研究首先在健康个体(BH01样本)中评估了FDI在基因组不同功能区域的表现,发现FDI在染色质开放区域和转录起始位点(TSS)附近显著升高,呈现出与DNase-seq信号高度一致的空间分布特征。进一步结合外周血单核细胞(PBMCs)的公开表达数据进行分析,发现FDI在TSS处的分布还与基因表达水平呈正相关,说明其对转录活跃区域的开放状态具有良好刻画能力。上述结果表明,FDI能够准确捕捉cfDNA中蕴含的染色质可及性信息。
二、片段离散度可以挖掘染色质开放区域并增强癌症早期诊断能力
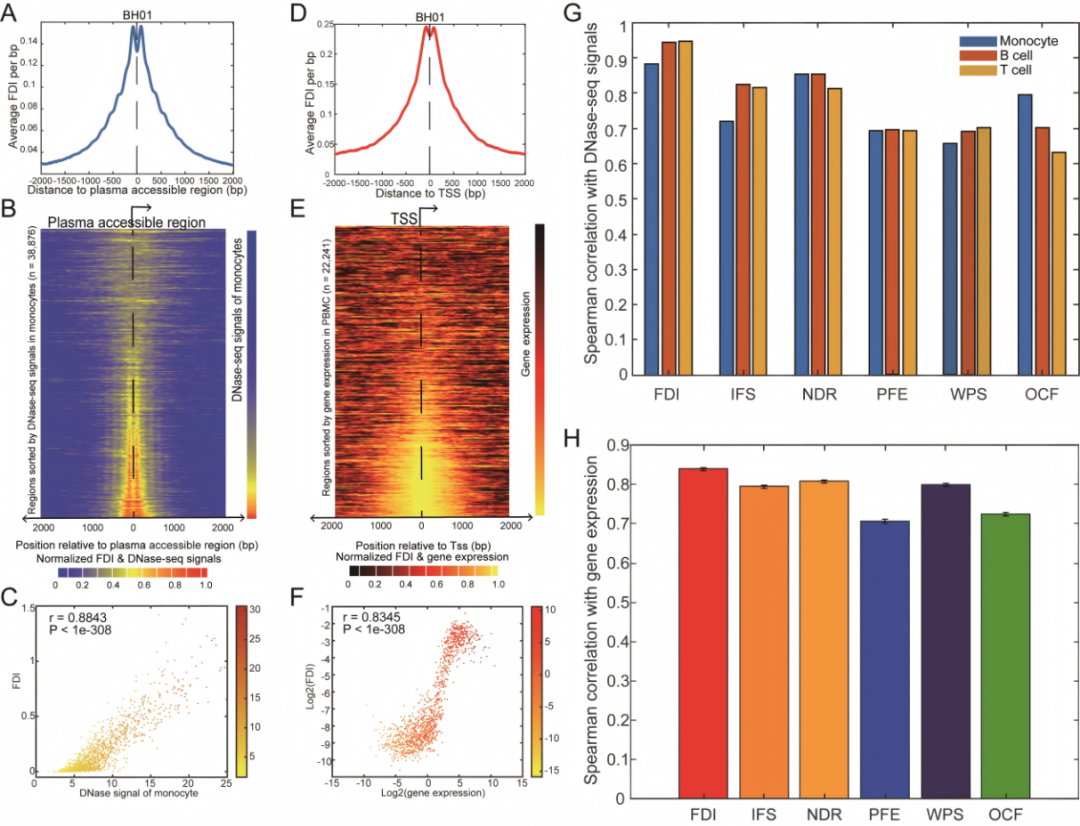
在确认FDI能够反映染色质可及性之后,作者进一步开发了一套基于FDI的统计分析方法,用于识别基因组中具有显著片段离散度特征的区域。这些“高离散区域”在空间分布上与开放染色质高度一致,富集启动子、增强子等功能元件,提示其在反映功能活性方面具有潜在价值。
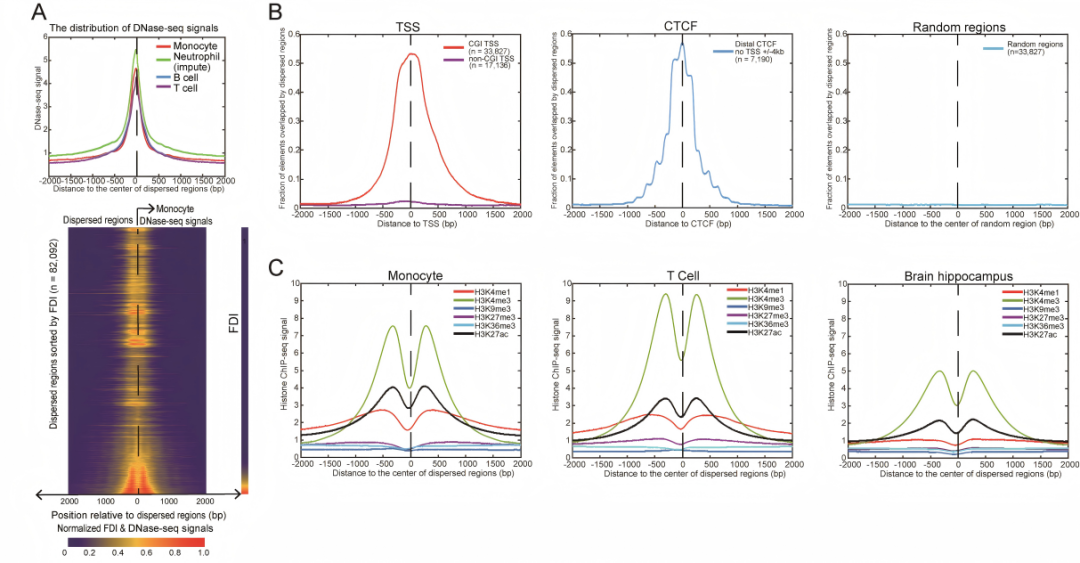
基于这些高离散区域,作者构建了FDI-Oncology-Model,提取其FDI特征作为输入,用于区分癌症患者与健康个体。在多个独立癌症队列中的评估结果显示,该模型具有稳健的早期诊断性能,验证了片段离散度在液体活检中的应用潜力。
三、FDI-Oncology-Model展现稳健诊断能力并揭示潜在机制
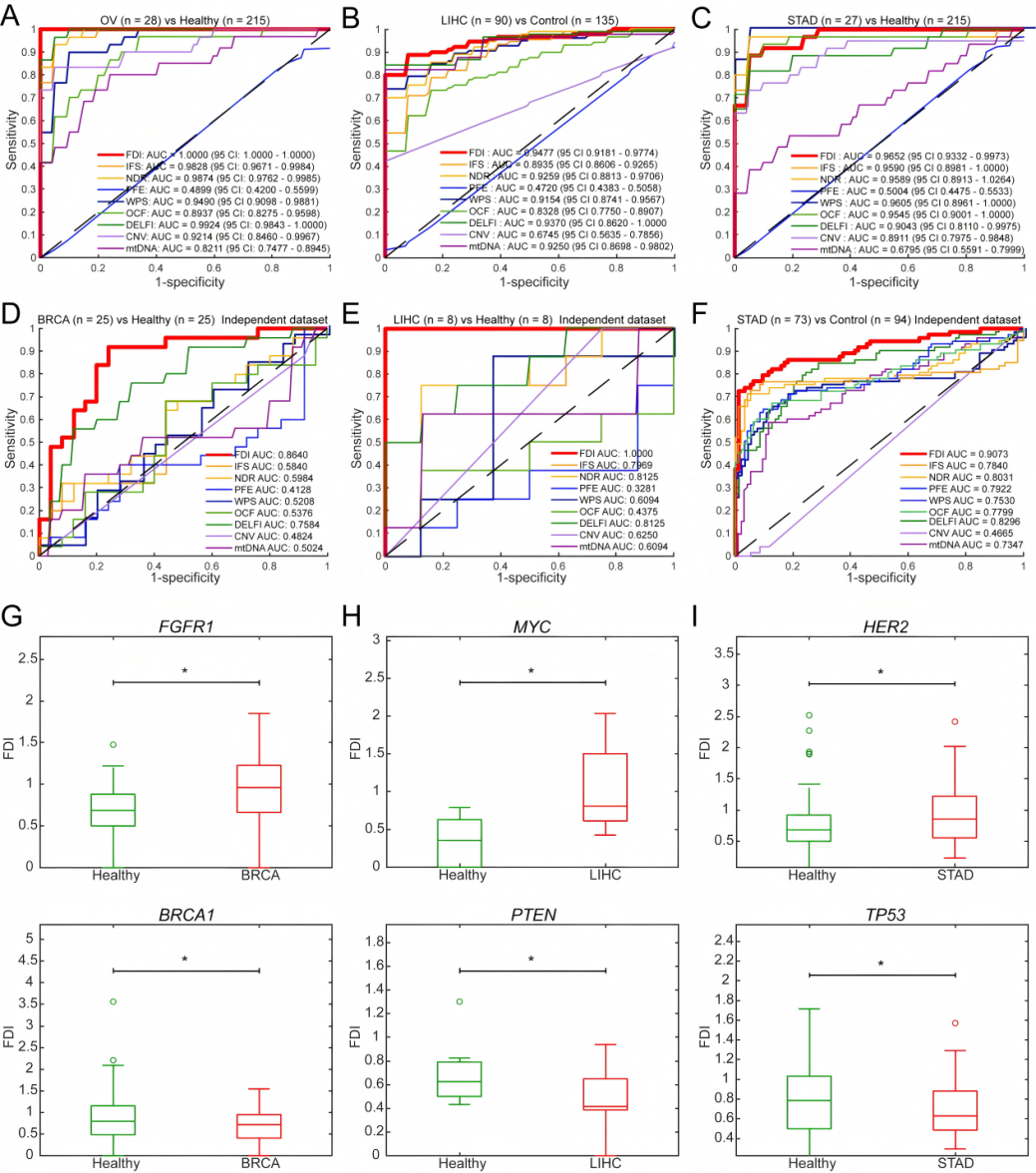
为了进一步评估FDI在真实临床场景中的表现,作者使用多个包含良性病变和炎症样本的独立数据集,系统分析了FDI-Oncology-Model的诊断能力。结果显示,FDI分数在癌症与非癌症样本之间存在显著差异,但在健康人群与良性结节、炎症、肝硬化等非癌状态之间则无明显差别,体现出良好的特异性。作者还尝试基于FDI进行癌症患者的预后分层,发现FDI-Oncology-Model得分高低与肺癌患者的生存结局存在统计学差异。进一步的功能注释和motif富集分析表明,这些离散区域富集了与癌症相关的通路和转录因子结合位点,提示FDI在具备诊断性能的同时,也能够提供一定的生物学解释力,为临床在结果解读与风险评估上带来更丰富的信息支持。
研究支持
华中农大章文教授/周雄辉副教授,军事医学科学院陈河兵副研究员为该论文的通讯作者,博士生王运泽为该文的第一作者,博士生熊展坤、宗湛翔,硕士生侯钰莹、卢立波和本科生王博参与了该研究。
论文链接
https://www.cell.com/cell-reports-methods/fulltext/S2667-2375(25)00119-5
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言


















#cfDNA# #片段离散度#
23 举报